A Pandemia de COVID-19 nos munic´ıpios do Estado do Paran´a
uma investigac¸˜ao via An´alise de Agrupamento
Palavras-chave:
Análise de agrupamento, Clusters, J48, Naive Bayes, Covid-19Resumo
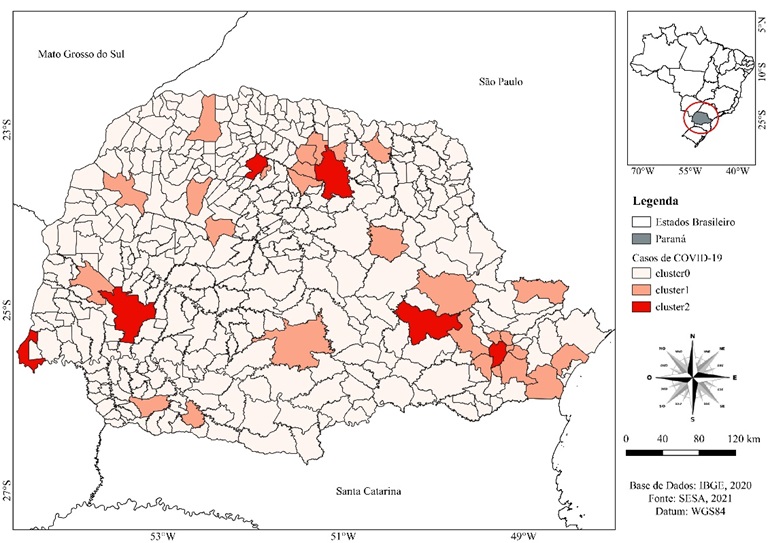
A presente pesquisa aborda o contexto epidemiológico da pandemia de COVID-19, focando o panorama específico do Brasil, com foco especial no Estado do Paraná, aproximadamente um ano após a implementação do primeiro confinamento. O objetivo geral desta pesquisa consiste em aplicar a técnica de análise de agrupamento conhecida como k-means para categorizar os municípios paranaenses com base em duas variáveis principais: o número de casos confirmados eon ´úmero de ´obitos por COVID-19. Para alcan¸car esse prop´osito, foram utilizados dados fornecidos pela Secretaria de Saúde do Paraná, abrangendo o período de 1º de janeiro de 2021 a 15 de março de 2021. Os resultados obtidos revelaram a identificação ¸c˜ao de três clusters que se destacaram como ´otimos, evidenciando divergências nos padrões de incidência de casos e ´óbitos entre os municípios. Notavelmente, observou-se uma correlação entre a densidade populacional e a frequência de casos e ´óbitos, com ´áreas mais densamente povoadas tendendo a registrar números mais elevados. Além disso, a avaliação da precisão dos algoritmos J48 e Naive Bayes na classificação dos clusters apresentou resultados satisfatórios. Consequentemente, conclui-se
que a técnica de agrupamento de empregados revelou-se eficaz na identificação de semelhanças nos padrões de propagação da COVID-19, oferecendo evidências relevantes para a formulação de Estratégias direcionadas e eficientes no enfrentamento da pandemia, especialmente nas regiões mais impactadas.
Referências
ALVES, H. J. P.; FERNANDES, F. A.; LIMA, K. P.; BATISTA, B. D. O.; FERNANDES, T. J. A pandemia da COVID-19 no Brasil: uma aplicação do método de clusterização k-means. Research, Society and Development, v. 9, n. 10, 2020. DOI: http://dx.doi.org/10.33448/rsd-v9i10.9059.
CHARRAD, M., GHAZZALI, N., BOITEAU, V., NIKNAFS, A. Determining the best number of clusters in a data set. Package NbClust, 2015. Recuperado de http://cran.rediris.es/ web/packages/NbClust/NbClust.pdf.
EMAMI, A.; JAVANMARDI, F.; PIRBONYEH, N.; AKBARI, A. Prevalence of Underlying Diseases in Hospitalized Patients with COVID-19: a Systematic Review and Meta-Analysis. Arch Acad Emerg Med. 8(1): e35, mar, 2020.
FAVERO, L. P.; BELFIORE, P. Data Science for Business and Decision Making. Academic Press, Cambridge, MA, USA, 2019
FAYYAD, U.M. et al. Advances in knowledge discovery and data mining. Massachusetts: AAAI Press, 1996.
GUIMARAES, R. M.; ELEUTERIO, T. D. A.; MONTEIRO-DA-SILVA, J. H. C. Estratificação de risco para predição de disseminação e gravidade da Covid-19 no Brasil. Revista Brasileira De Estudos De População, 37, 1-17, 2020. DOI:
http://dx.doi.org/10.20947/s0102- 3098a0122.
IRITANI, O.; OKUNO, T.; HAMA, D.; KANE, A.; KODERA, K.; MORIGAKI, K.; TERAI, T.; MAENO, N.; MORIMOTO, S. Clusters of covid-19 in long-term care hospitals and facilities in japan from 16 january to 9 may 2020. Geriatrics & gerontology international, 20(7), 715-719, 2020. DOI: 10.1111/ggi.13973.
JAMES, N.; MENZIES, M. Cluster-based dual evolution for multivariate time series: Analyzing covid-19. Chaos: An Interdisciplinary Journal of Nonlinear Science, 30, 2020. DOI: https://doi.org/10.1063/5.0013156.
LU, R.; ZHAO, X.; LI, J.; NIU, P.; YANG, B.; WU, H.; WANG, W. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. The Lancet. v.395, Feb 22, P. 565-574, 2020.
https://doi.org/10.1016/S0140-6736(19)33096-X.
MACIEL, E. L.; JABOR, P.; GONC¸ ALVES JUNIOR, E.; TRISTÃO-SÁ, R.; LIMA, R.C.D.; REIS-SANTOS, B.; LIRA, P.; BUSSINGUER, E. C. A.; ZANDONADE, E. Fatores associados ao óbito hospitalar por covid-19 no Espírito Santo. Epidemiologia e Serviços de Saúde, 29(4), 1-11, 2020. DOI: 10.5123/S1679-49742020000400022.
R CORE TEAM. R: A language and environment for statistical computing. R Foundation for Statistical computing, Vienna, 2020. Dispon´ıvel em: https://www.Rproject.org/.
RATKOWSKY, D.; LANCE, G. Criterion for determining the number of groups in a classification. Australian Computer Journal, 10(3), 115-117, 1978.
SESA, Secretaria da saúde: Informe Epidemiológico Coronavírus (COVID-19). Boletim epidemiológico, Curitiba, Março, 2021. Dispon ́ıvel em:
https://www.saude.pr.gov.br/sites/default/arquivos restritos/files/documento/2021-03/informe epidemiológico 15 03 2021.pdf.
WITTEN IH, F. Data Mining: Practical Machine Learning Tools and Techniques. 2nd edition. Morgan Kaufmann, San Francisco, 2005.
Downloads
Publicado
Como Citar
Edição
Seção
Licença
Proposta de Política para Periódicos de Acesso Livre
Autores que publicam nesta revista concordam com os seguintes termos:
- Autores mantém os direitos autorais e concedem à revista o direito de primeira publicação, com o trabalho simultaneamente licenciado sob a Licença Creative Commons Attribution que permite o compartilhamento do trabalho com reconhecimento da autoria e publicação inicial nesta revista.
- Autores têm autorização para assumir contratos adicionais separadamente, para distribuição não-exclusiva da versão do trabalho publicada nesta revista (ex.: publicar em repositório institucional ou como capítulo de livro), com reconhecimento de autoria e publicação inicial nesta revista.
- Autores têm permissão e são estimulados a publicar e distribuir seu trabalho online (ex.: em repositórios institucionais ou na sua página pessoal) a qualquer ponto antes ou durante o processo editorial, já que isso pode gerar alterações produtivas, bem como aumentar o impacto e a citação do trabalho publicado (Veja O Efeito do Acesso Livre).
